A developmental regulator of bacterial division
Agência FAPESP, 8th April 2015
Karina Toledo
Available only in Portuguese
Estudo da USP desvenda mecanismo de inibição da divisão bacteriana
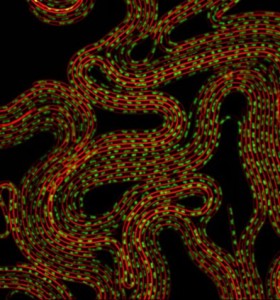
Células de Bacillus subtilis forçadas a produzir a proteína MciZ não conseguem se dividir e se transformam em longos filamentos multinucleados. A cor vermelha representa a membrana celular e a verde, o DNA bacteriano (divulgação)
Em um artigo divulgado segunda-feira (06/4) na revista Proceedings of the National Academy of Sciences, pesquisadores do Instituto de Química da Universidade de São Paulo (IQ-USP) descreveram em detalhes o mecanismo pelo qual uma proteína chamada MciZ inibe o processo de divisão bacteriana.
A investigação foi conduzida no âmbito do projeto “Smolbnet 2.0: combinando genética e RMN para dissecar interações proteína-proteína fundamentais para o funcionamento do complexo de divisão bacteriana”, coordenado por Frederico José Gueiros Filho e apoiado pela FAPESP.
“O estudo de proteínas que naturalmente inibem a divisão de bactérias, além de permitir que compreendamos como o processo de divisão é regulado pela célula, pode inspirar o desenvolvimento de inibidores sintéticos e abrir caminho para uma nova classe de antibióticos. As proteínas que compõem o aparato de divisão celular são um alvo terapêutico atraente que ainda não é atacado pelos antibióticos disponíveis comercialmente”, afirmou Gueiros à Agência FAPESP.
De acordo com o pesquisador, existem cerca de 20 proteínas especializadas em executar a divisão das bactérias, sendo a FtsZ a mais importante de todas. Para estudar a interação entre essas proteínas, o grupo do IQ-USP usa como modelo a espécie Bacillus subtilis, que costuma habitar o solo e não causa doença em humanos.
“É um organismo muito fácil de ser estudado e, portanto, serve de exemplo para outros. Como o mecanismo de divisão é semelhante em todas as bactérias – sempre dependente de FtsZ –, o que aprendemos com Bacillus subtilis é aplicável a muitas outras espécies, inclusive as patogênicas”, disse.
Quando tem início o processo de reprodução bacteriana, explicou o pesquisador, as moléculas de FtsZ se entrelaçam umas às outras formando filamentos. Esses filamentos se agrupam em uma espécie de anel contrátil, que marca o local onde ocorrerá a divisão.
Ao formar essa “cintura” na parte interna, a FtsZ atrai outras proteínas de divisão para o anel, que alteram a parede externa da bactéria. No momento da divisão, a parede bacteriana para de se expandir no comprimento e passa a crescer para dentro, formando um septo que separa a bactéria em duas células-filhas.
Mas para evitar que a divisão ocorra na hora ou local inadequados, existem no interior da célula proteínas que funcionam como inibidores naturais, capazes de impedir a formação do anel contrátil. Entre as mais importantes estão a MciZ e a MinC – alvos do projeto coordenado por Gueiros.
“Nós já sabíamos que a MciZ se ligava à FtsZ e isso de alguma maneira impedia a formação do anel contrátil. Mas para entender como isso ocorre em nível molecular recorremos a técnicas de biologia estrutural”, contou o pesquisador.
Por meio de um método conhecido como cristalografia de raios X, os pesquisadores desvendaram a estrutura tridimensional do complexo formado pelas proteínas FtsZ e MciZ. Já com a ajuda da ressonância magnética nuclear (RMN), os cientistas estudaram a estrutura tridimensional da MciZ isolada.
O trabalho contou com a colaboração das pesquisadoras Ana Carolina Zeri, do Laboratório Nacional de Biociências (LNBio), e Andrea Dessen, que atua no Instituto de Biologia Estrutural (IBS) em Grenoble, na França, e também no LNBio por meio do programa São Paulo Excellence Chair (SPEC), da FAPESP.
Alvo terapêutico
“Quando desvendamos a estrutura de complexos proteicos, podemos descobrir que porções de cada uma das proteínas estão se tocando e, dessa forma, deduzir o mecanismo pelo qual uma está inibindo a outra. O sítio onde ocorre a interação é um alvo para o desenvolvimento de fármacos”, disse Gueiros.
Uma série de experimentos bioquímicos in vitro foi realizada para complementar o entendimento da interação entre as duas proteínas. Os cientistas observaram que a molécula de MciZ se liga na ponta dos filamentos, exatamente onde deveria entrar uma outra molécula de FtsZ. Dessa forma, o filamento para de crescer.
“Na ausência de MciZ, o filamento composto de moléculas de FtsZ cresce até atingir cerca de 40 subunidades e então ocorre a formação do anel contrátil. Mas basta uma pequena quantidade de MciZ – algo na proporção de 1 para 20 – para reduzir o tamanho do filamento pela metade e tornar impossível a formação do anel e a divisão celular”, explicou Gueiros.
Segundo o pesquisador, trata-se de um mecanismo de inibição poderoso, pois não é necessário que a MciZ se ligue a cada uma das subunidades de FtsZ. “Isso deve ser levado em conta na hora de desenhar inibidores sintéticos, pois permite que o fármaco atue em concentrações mais baixas”, avaliou.
Em um trabalho anterior divulgado na revista PLoS One em 2013, o grupo investigou mutações na cadeia de aminoácidos que formam a proteína FtsZ e a impedem de se ligar à MinC, também inibidora da divisão.
“O mecanismo de atuação nesse caso é diferente, pois a MinC não se liga na ponta do filamento e sim na lateral da molécula de FtsZ. Ainda assim, ela impede a formação do anel contrátil”, contou Gueiros.
Apesar das potenciais implicações práticas dos resultados obtidos, a principal motivação do grupo de Gueiros é compreender os mecanismos celulares em seu nível mais fundamental.
“A boa pesquisa básica, além de avançar a nossa compreensão da natureza, tem sido a principal fonte das descobertas que alimentam a inovação e a tecnologia”, disse o pesquisador.
O artigo FtsZ filament capping by MciZ, a developmental regulator of bacterial division (doi: 10.1073/pnas.1414242112) pode ser lido em www.pnas.org/content/early/2015/04/02/1414242112.abstract” target=.
